Un trabajo del CIBERES publicado en Microbiology Spectrumo ofrece nuevas claves de la adaptación fisiológica y metabólica de bacterias causantes de infección respiratoria.
 Las vías bajas del sistema respiratorio humano, y en particular los pulmones de pacientes que sufren patologías respiratorias crónicas como la Enfermedad Pulmonar Obstructiva Crónica (EPOC), están colonizadas por patógenos bacterianos que desarrollan estrategias de adaptación sofisticadas para evadir la inmunidad respiratoria. Entre otros, estos patógenos desarrollan estrategias destinadas a superar la restricción de nutrientes impuesta por el sistema respiratorio para mitigar la patogenicidad durante procesos infecciosos.
Las vías bajas del sistema respiratorio humano, y en particular los pulmones de pacientes que sufren patologías respiratorias crónicas como la Enfermedad Pulmonar Obstructiva Crónica (EPOC), están colonizadas por patógenos bacterianos que desarrollan estrategias de adaptación sofisticadas para evadir la inmunidad respiratoria. Entre otros, estos patógenos desarrollan estrategias destinadas a superar la restricción de nutrientes impuesta por el sistema respiratorio para mitigar la patogenicidad durante procesos infecciosos.
Según la Organización Mundial de la Salud, la EPOC es la tercera causa más frecuente de muerte a nivel mundial, por lo que entender las claves fisiológicas de adaptación de patógenos bacterianos que causan infección pulmonar en estos pacientes es clave para dirigir nuevos desarrollos terapéuticos.
El trabajo liderado por Junkal Garmendia, investigadora del Instituto de Agrobiotecnología (IdAB-CSIC) y jefa de grupo del CIBERES, es pionero en la identificación de claves fisiológicas y metabólicas de adaptación de la bacteria Haemophilus influenzae durante un proceso de infección respiratoria. H. influenzae forma parte de la microbiota respiratoria humana en vías altas, y es también un patógeno oportunista causante de infección pulmonar en pacientes inmunocomprometidos, como aquellos que sufren Enfermedad Pulmonar Obstructiva Crónica (EPOC).
Nuevos avances en la investigación
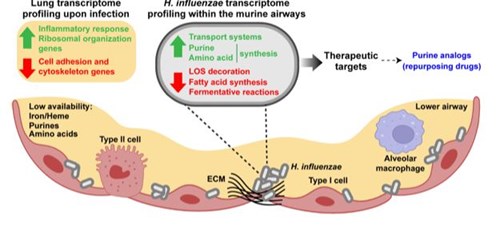
Ahora, el trabajo liderado por la Dra. Garmendia, y realizado en colaboración con investigadores de la Universidad de West Virginia (EEUU), va un paso más allá, y caracteriza la reprogramación transcripcional a nivel de genoma completo (reprogramación de transcriptoma) que experimenta H. influenzae durante un proceso de infección respiratoria. Para ello, los investigadores utilizaron secuenciación de transcriptoma in vivo (in vivo RNA-seq) para analizar los cambios que ocurren en el perfil transcriptómico de este patógeno durante un proceso de infección respiratoria en un modelo murino disponible y previamente caracterizado.
El análisis transcriptómico de las bacterias recuperadas de muestras de lavado broncoalveolar procedentes de animales infectados mostró una clara reprogramación metabólica durante la infección.
En concreto, se observó una importante sobre-expresión de los genes que codifican las enzimas implicadas en la ruta de síntesis de purinas de novo, así como de genes que codifican proteínas responsables de la síntesis de amino ácidos no aromáticos, y de parte de la maquinaria de competencia natural de H. influenzae.
Por el contrario, los investigadores observaron una clara reducción en la expresión de genes que codifican enzimas implicadas en la síntesis de ácidos grasos, y en la síntesis de peptidoglicano y lipooligosacárido, que son elementos clave en la arquitectura de la pared bacteriana.
El análisis in vivo RNA-seq realizado resultó ser una excelente herramienta de escrutinio de nuevas dianas terapéuticas, ya que la inactivación de genes bacterianos sobre-expresados durante la infección ocasionó atenuación in vivo, estableciendo una correlación entre sobre-expresión de genes implicados en la síntesis de purinas y su contribución al fitness del patógeno durante la infección. El potencial terapéutico de la inhibición de la síntesis de purinas de H. influenzae fue además confirmado mediante el uso de dos análogos de purinas, que mostraron efecto antibacteriano como fármacos de reposicionamiento.
En conjunto, “este trabajo pone de manifiesto el papel activo del metabolismo bacteriano en la capacidad pato-adaptativa de H. influenzae durante la infección respiratoria, identifica la síntesis de purinas como posible diana terapéutica, y muestra el incuestionable potencial de las metodologías –ómicas aplicadas a modelos de trabajo in vivo como poderosas herramientas de escrutinio de dianas para futuros desarrollos terapéuticos en la lucha frente a procesos infecciosos respiratorios de gran relevancia clínica” concluye la Dra. Garmendia. Y agradece "la donación realizada por la familia de Mª Araceli Lavela González, recibida a través de la Asociación APEPOC (Asociación de Pacientes con EPOC). Trabajamos para generar conocimiento que permita implementar mejoras en el diagnóstico, pronóstico y tratamiento de la EPOC y sus exacerbaciones asociadas"
Trabajo publicado en:
Euba B, Gil-Campillo C, Asensio-López J, López-López N, Sen-Kilic E, Díez-Martínez R, Burgui S, Barbier M, Garmendia J. In vivo genome-wide gene expression profiling reveals that Haemophilus influenzae purine synthesis pathway benefits its infectivity within the airways. Microbiol Spectr. 2023 May 17:e0082323. doi: 10.1128/spectrum.00823-23. 7:e0082323. doi: 10.1128/spectrum.00823-23.
https://www.ciberes.org/noticias/el-metabolismo-bacteriano-una-fuente-de-dianas-terapeuticas-crucial-en-el-desarrollo-de-antimicrobianos-frente-a-patogenos-respiratorios
 Actualidad
Actualidad  El metabolismo bacteriano, una fuente de dianas terapéuticas crucial en el desarrollo de antimicrobianos frente a patógenos respiratorios
El metabolismo bacteriano, una fuente de dianas terapéuticas crucial en el desarrollo de antimicrobianos frente a patógenos respiratorios Actualidad
Actualidad  El metabolismo bacteriano, una fuente de dianas terapéuticas crucial en el desarrollo de antimicrobianos frente a patógenos respiratorios
El metabolismo bacteriano, una fuente de dianas terapéuticas crucial en el desarrollo de antimicrobianos frente a patógenos respiratorios Las vías bajas del sistema respiratorio humano, y en particular los pulmones de pacientes que sufren patologías respiratorias crónicas como la Enfermedad Pulmonar Obstructiva Crónica (EPOC), están colonizadas por patógenos bacterianos que desarrollan estrategias de adaptación sofisticadas para evadir la inmunidad respiratoria. Entre otros, estos patógenos desarrollan estrategias destinadas a superar la restricción de nutrientes impuesta por el sistema respiratorio para mitigar la patogenicidad durante procesos infecciosos.
Las vías bajas del sistema respiratorio humano, y en particular los pulmones de pacientes que sufren patologías respiratorias crónicas como la Enfermedad Pulmonar Obstructiva Crónica (EPOC), están colonizadas por patógenos bacterianos que desarrollan estrategias de adaptación sofisticadas para evadir la inmunidad respiratoria. Entre otros, estos patógenos desarrollan estrategias destinadas a superar la restricción de nutrientes impuesta por el sistema respiratorio para mitigar la patogenicidad durante procesos infecciosos.